Coding as a Service:
Librería pypsdier, aprendizajes y metodología.
Sebastián Flores
Pycon Argentina 2020

Coding as a Service: librería pypsdier, aprendizajes y metodología.¶
Observaciones:¶
- @sebastiandres en twitter y github.
- Opiniones y críticas a título personal.
- No se necesita conocer de química, matemática o python avanzado.
- Presentación disponible en https://www.github.com/sebastiandres/charlas y realizada con jupyter+RISE.
Coding as a Service: librería pypsdier, aprendizajes y metodología.¶
Autores¶

Sebastián Flores
M.Sc. Mathematical and Computational EngineeringM.Sc. Mécanique Numérique
Ingeniero Civil Matemático
u-planner - Chile

Pedro Valencia
Ph.D Ingeniería BioquímicaM.Sc. Ingeniería Bioquímica
Bioquímico
UTFSM - Chile
- Primeras implementaciones en MATLAB:
- Problema: Cada nueva reacción química requiere una nueva implementación numérica.
- Repetición de código y mantención tediosa.
- Desarrollo en Python:
- Código genérico: librería pypsdier.
- Interface simple e instalación por pypi.
- Reacciones químicas definidas como funciones.
- Número arbitrario de sustancias (sustratos o productos).
- Tamaño de partículas definidas por una lista de radios y frecuencias.
- Impacto:
- 9 publicaciones.
- Código abierto y en constante evolución.
¿Cómo colaborar de manera eficiente?
El problema más general:
¿Cuál es la manera más sencilla de entregar "Coding As A Service"?
Tiene que ser tan fácil que puedas explicárselo a tu abuela.

Coding as a Service: librería pypsdier, aprendizajes y metodología.¶
Nuestro problema a resolver¶
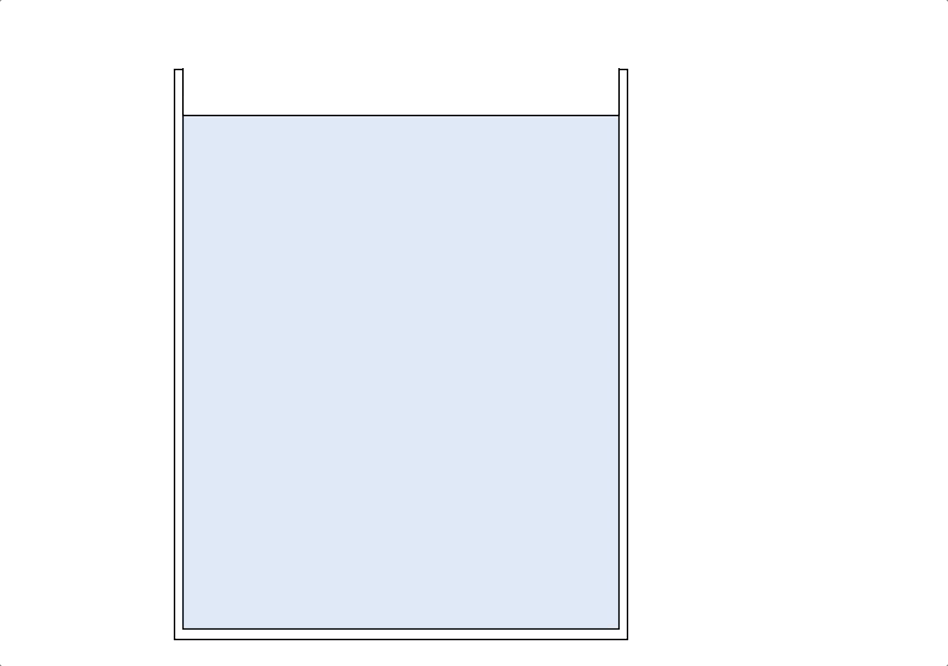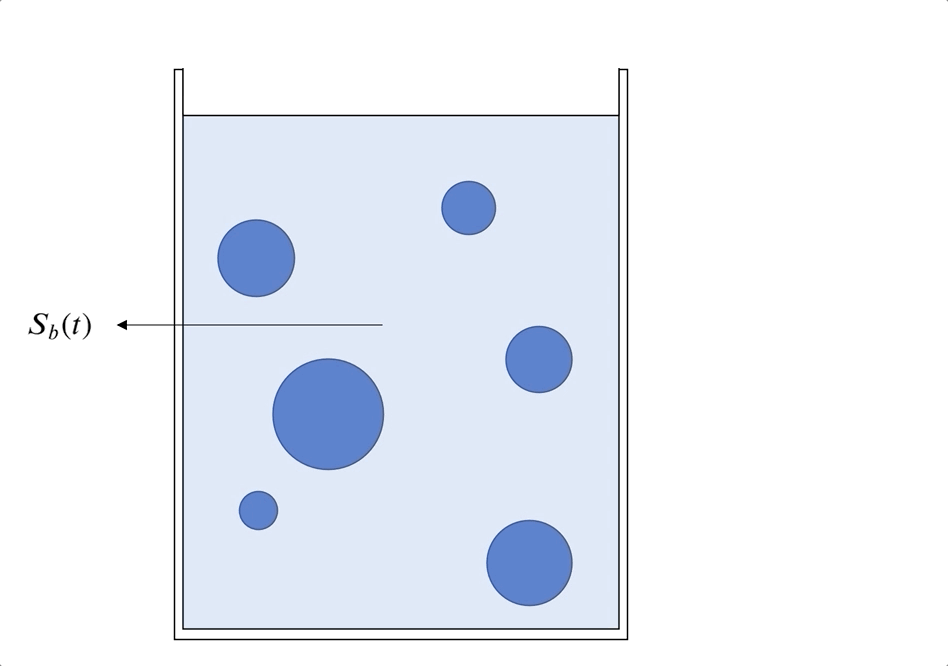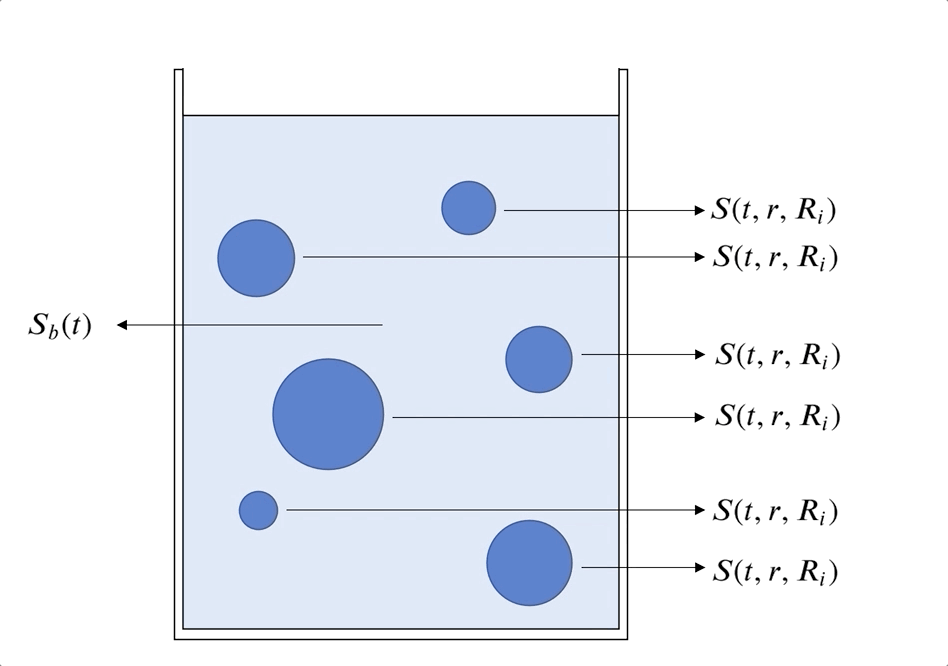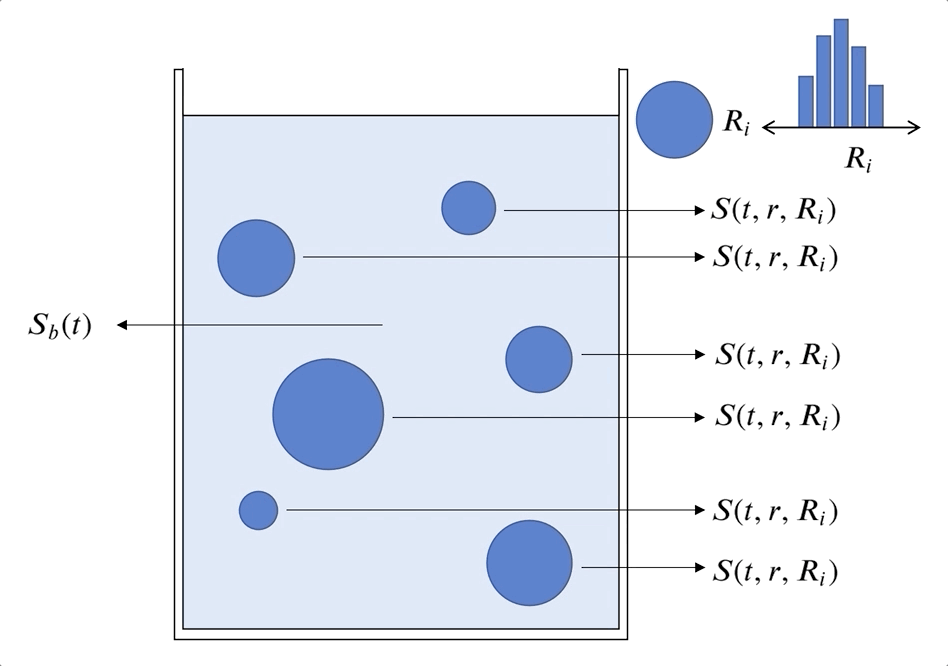
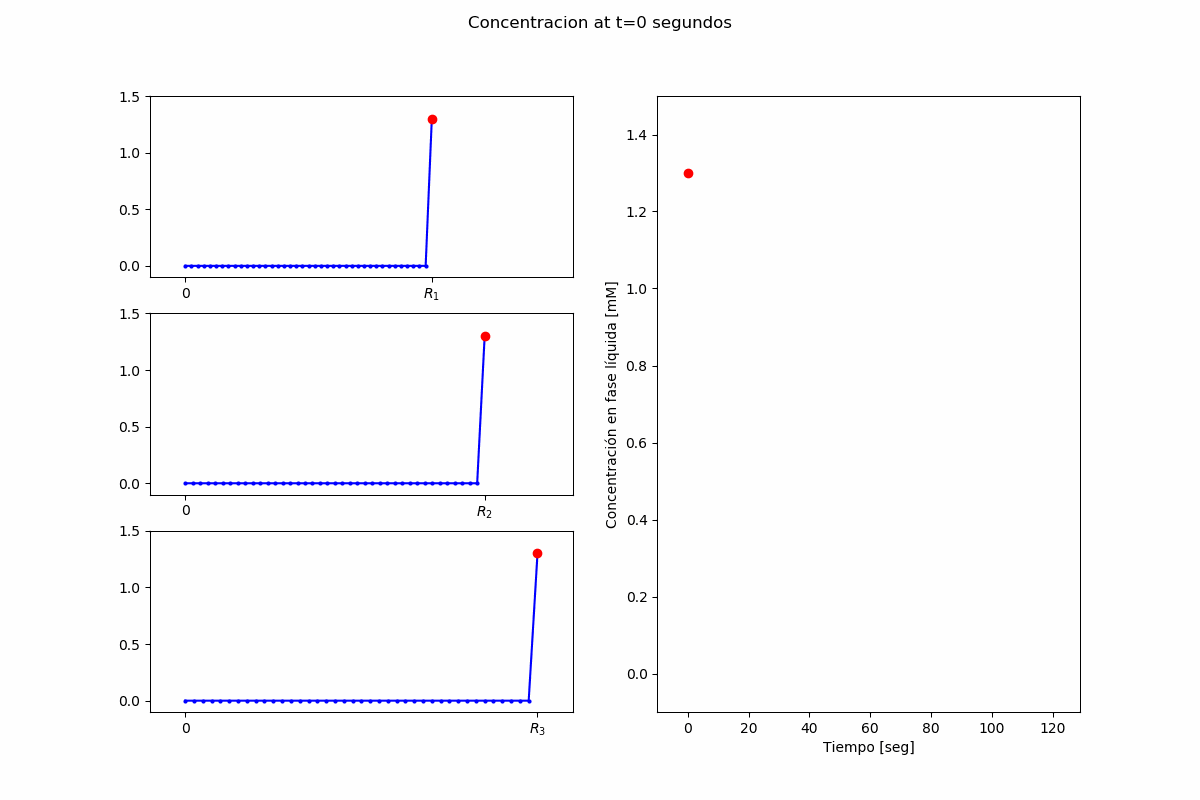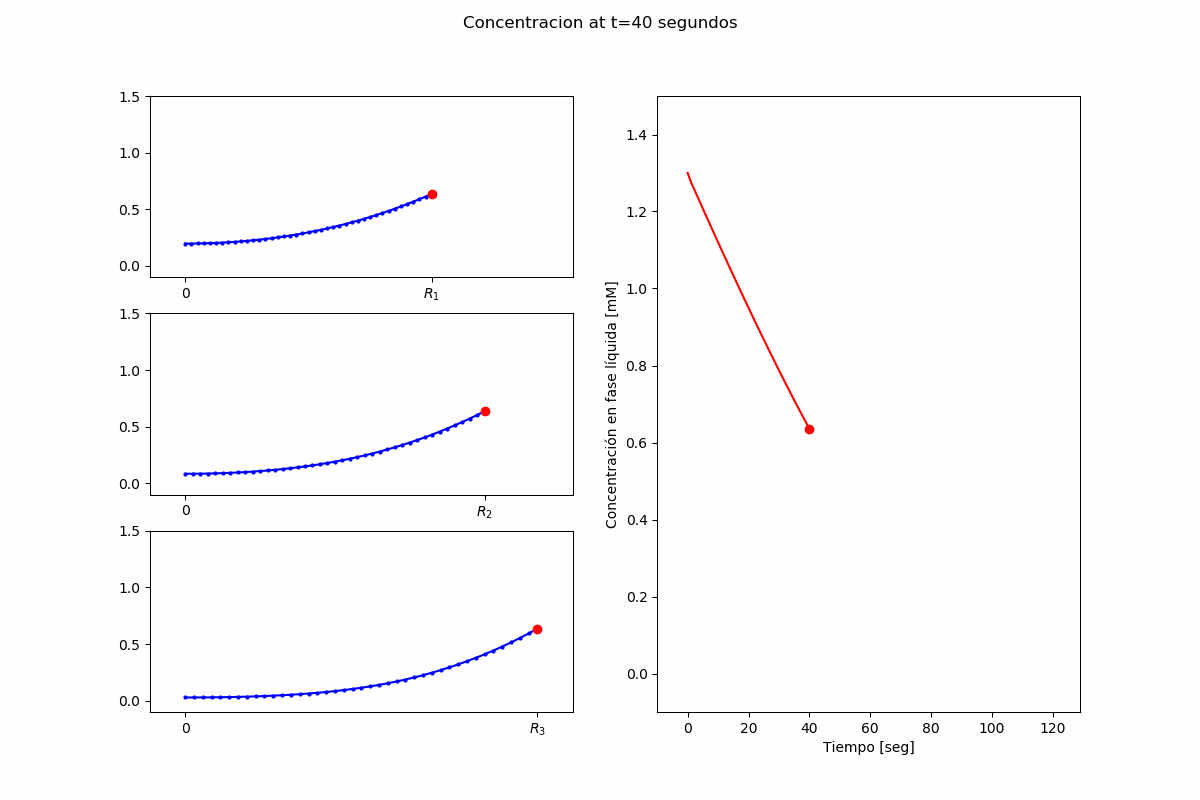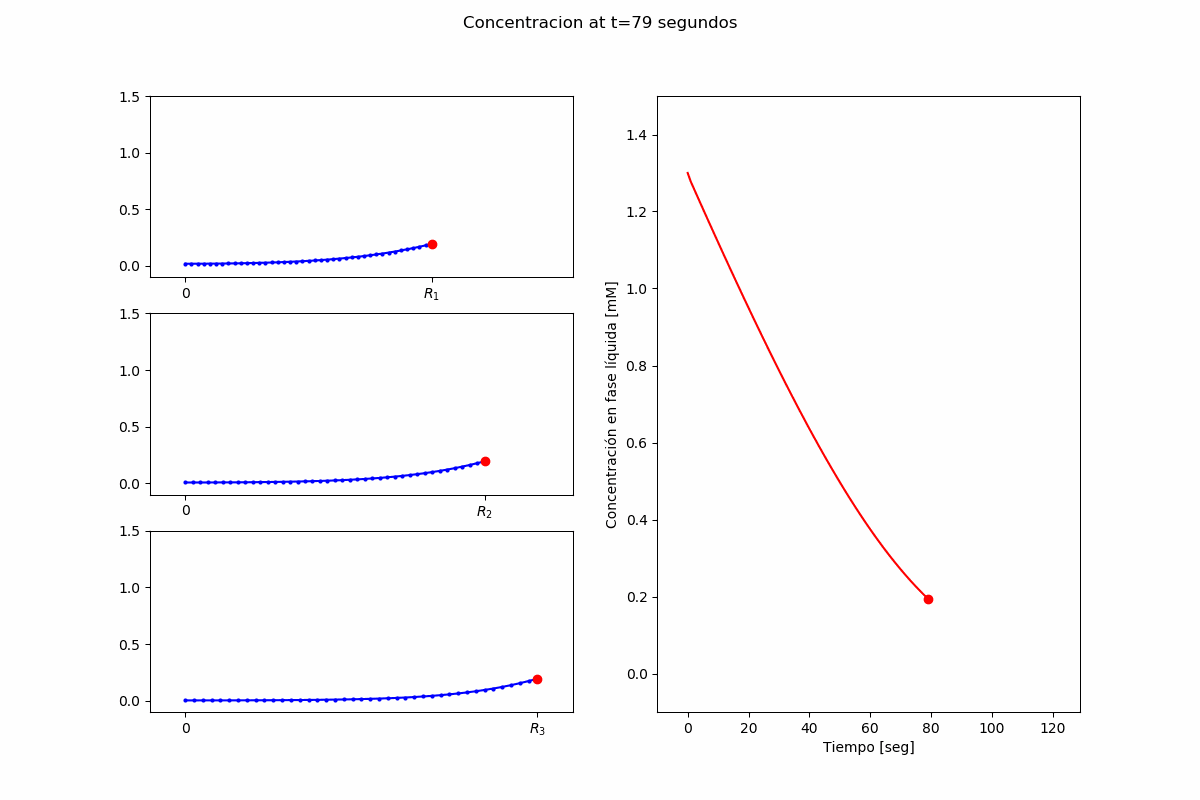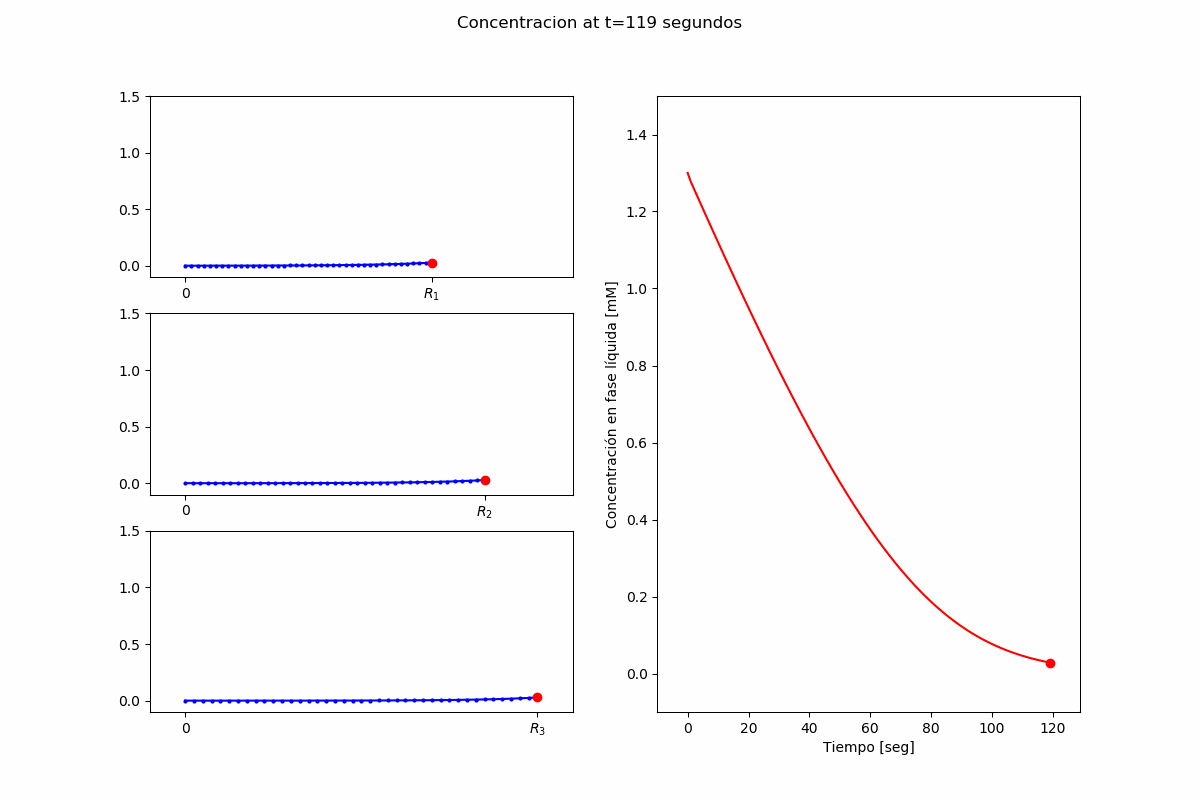
El problema es obtener $S_b(t)$ y $S(t,r,R_i)$, para $t>0$, $0<r<R_i$.
$$ \frac{\partial S}{\partial t}(t,r,R_i) = D_S \left(\frac{\partial^2 S}{\partial r^2}(t,r,R_i) + \frac{2}{r}\frac{\partial S}{\partial r}(t,r,R_i)\right) - V_e\left(S, r, t \right) $$Condición de borde en el centro de las partículas:
\begin{align} \frac{\partial S}{\partial r}(t, 0, R_i) = 0 \textrm{ para } t > 0 \end{align}Condiciones de borde en la superficie de la partícula:
\begin{align} S_b(t) = S(t, R_i,R_i) \textrm{ para } t > 0 \end{align}\begin{align} \frac{d S_b}{d t}(t) &= - 3 D_S \frac{V_c}{V_R E \left[ R^3 \right] } E \left[ R^2 \left. \frac{\partial S}{\partial r} \right|_{r=R} \right] \textrm{ para } t > 0 \end{align}Con la condición inicial
\begin{align} S_b(0) &= S_0 \\ S(0,r, R_i) &= 0 \textrm{ para } 0 \leq r < R_i \end{align}Esta es la parte tiene solución directa y conocida:
- Discretizar ecuaciones utilizando diferencias finitas para el tiempo ($\Delta t$) y espacio ($\Delta R_i$).
- Asegurar estabilidad numérica dependiendo de los inputs.
- Implementación flexible con python + numpy + matplotlib + otros.
Coding as a Service: librería pypsdier, aprendizajes y metodología.¶
Los Desafíos¶
Desafíos organizacionales:
- ¿Cómo colaborar?
- ¿Cómo distribuir el código?
- ¿Cómo hacer fácil el uso?
- ¿Cómo hacer reproducible las simulaciones?
- ¿Cómo documentar el código?
- ¿Cómo almacenar los resultados de las simulaciones?
Esta parte no tiene una solución directa:
- No había mucha información al respecto.
- Cada equipo lo resuelve a su manera.
- Existen muchas herramientas que nos simplifican la vida.
Coding as a Service: librería pypsdier, aprendizajes y metodología¶
Framework: ¿Qué necesitábamos?¶
(1) Propocionar instalación y versionamiento de python, jupyter y librerías necesarias.
(2) Facilitar desarrollo incremental considerando devs y users.
(3) Permitir una buena documentación para desarrolladores y usuarios.
(4) Exponer una interface simple al usuario final, permitiendo ocultar una implementación numérica compleja y con una curva de aprendizaje apropiado.
(5) Permitir reproducir, almacenar y compartir resultados de simulación.
(6) Permitir el uso de recursos computacionales en la nube.
Coding as a Service: librería pypsdier, aprendizajes y metodología¶
Necesidades¶
(4) Exponer una interface simple al usuario final, permitiendo ocultar una implementación numérica compleja y con una curva de aprendizaje apropiado.
(5) Permitir reproducir, almacenar y compartir resultados de simulación.
Solución¶
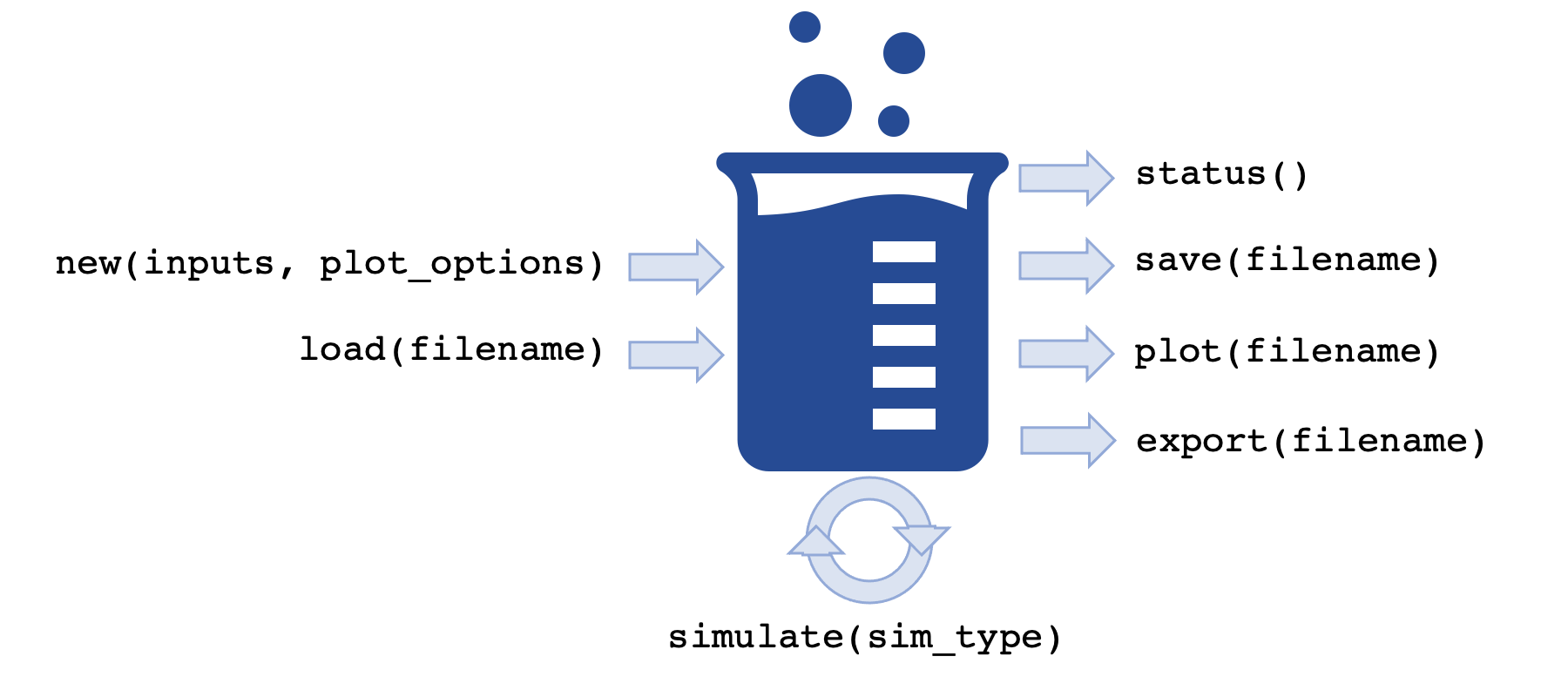
Solución¶
#!pip install pypsdier --upgrade
!pip install git+https://github.com/sebastiandres/pypsdier.git --upgrade
Collecting git+https://github.com/sebastiandres/pypsdier.git
Cloning https://github.com/sebastiandres/pypsdier.git to /private/var/folders/68/v1ds0ld152q_wk4sbcns3ckw0000gn/T/pip-req-build-mm52_b8o
Running command git clone -q https://github.com/sebastiandres/pypsdier.git /private/var/folders/68/v1ds0ld152q_wk4sbcns3ckw0000gn/T/pip-req-build-mm52_b8o
Building wheels for collected packages: pypsdier
Building wheel for pypsdier (setup.py) ... done
Created wheel for pypsdier: filename=pypsdier-1.1.1-cp37-none-any.whl size=14659 sha256=34556c2c7b1b3be6222355f403a45817e8b5a0154f44037f940a4923711fe71d
Stored in directory: /private/var/folders/68/v1ds0ld152q_wk4sbcns3ckw0000gn/T/pip-ephem-wheel-cache-csxbayor/wheels/57/71/0f/2f6f43ab73e6371b622a1e181130163a445c394d19b74a80d1
Successfully built pypsdier
Installing collected packages: pypsdier
Found existing installation: pypsdier 1.1.1
Uninstalling pypsdier-1.1.1:
Successfully uninstalled pypsdier-1.1.1
Successfully installed pypsdier-1.1.1
def MichaelisMenten(S, E0, k, K):
"""Definition for Michaelis Menten reaction with inputs E0 [mM], k [1/s] and K [mM]"""
return (-k*E0*S[0]/(K+S[0]), )
inputs = {}
inputs["SimulationTime"] = 30. # [s]
inputs["SavingTimeStep"] = 1. # [s]
inputs["CatalystVolume"] = 0.5 # [mL]
inputs["BulkVolume"] = 100.0 # [mL]
inputs["Names"] = ('Substrat',) # legend for the xls, reports and plots
inputs["InitialConcentrations"] = (1.3,) # [mM]
inputs["EffectiveDiffusionCoefficients"] = (5.3E-10,) # [m2/s]
inputs["CatalystParticleRadius"] = [40.0E-6, 60.0E-6, 80.0E-6] # [m]
inputs["CatalystParticleRadiusFrequency"] = [0.3, 0.5, 0.2] # []
inputs["ReactionFunction"] = MichaelisMenten # function
inputs["ReactionParameters"] = (41 , 0.13) # [1/s], [mM/s], parameters
inputs["CatalystEnzymeConcentration"] = 0.35 # [mM]
plot_options = {}
plot_options["label_x"] = "Tiempo de reacción [s]"
plot_options["label_y"] = "Concentración [mM]"
plot_options["title"] = "Simulación de Michaelis Menten para la PyconAr"
plot_options["ode_kwargs"] = {'label':'Enzima Libre', 'color':'blue', 'marker':'', 'markersize':6, 'linestyle':'dashed', 'linewidth':2}
plot_options["pde_kwargs"] = {'label':'Enzima Inmobilizada', 'color':'blue', 'marker':'', 'markersize':6, 'linestyle':'solid', 'linewidth':2}
plot_options["data_kwargs"] = {'label':'Experimento', 'color':'green', 'marker':'s', 'markersize':8, 'linestyle':'none', 'linewidth':2}
plot_options["data_x"] = [0.0, 30, 60, 90, 120]
plot_options["data_y"] = [1.3, 0.65, 0.25, 0.10, 0.0]
import pypsdier
SIM1 = pypsdier.SimulationInterface()
SIM1.new(inputs, plot_options)
SIM1.simulate("ode")
ODE: Solving without diffusional restriction (simplified problem). No porous particles are present. The substance and the enzyme are assumed to be diluted on the bulk phase Simulation completed.
SIM1.simulate("pde")
PDE: Solving the complete reaction-diffusion problem, considering a single substance and 3 particle radii. Simulated 030 secs out of 30 secs (Remaining time < 1 mins)
SIM1.save("pycon_saving_example.rde")
Saving simulation into file at /Users/sebastiandres/Desktop/Personales/github_repos/charlas/2020_11_XX_pycon_ar_pypsdier/pycon_saving_example.rde
SIM1.export_xls("pycon_excel_example.xls")
Saving simulation as xls file at /Users/sebastiandres/Desktop/Personales/github_repos/charlas/2020_11_XX_pycon_ar_pypsdier/pycon_excel_example.xls
SIM1.plot(figsize=(16,12))
import pypsdier
SIM2 = pypsdier.SimulationInterface()
SIM2.load("pycon_colab_example.rde") # load instead of new
Loaded a simulation from /Users/sebastiandres/Desktop/Personales/github_repos/charlas/2020_11_XX_pycon_ar_pypsdier/pycon_colab_example.rde
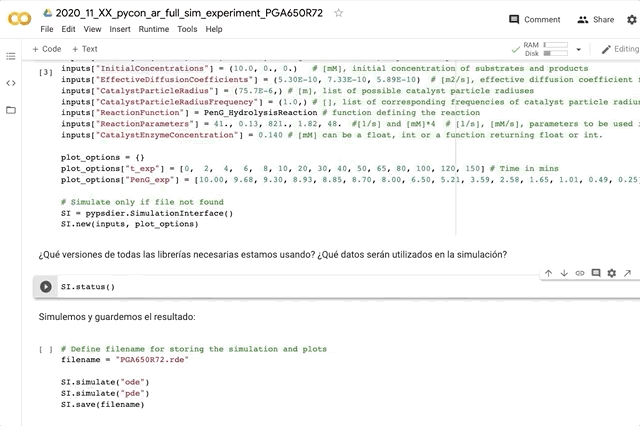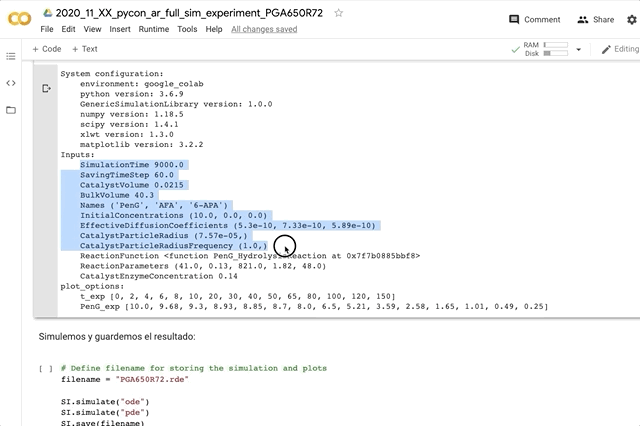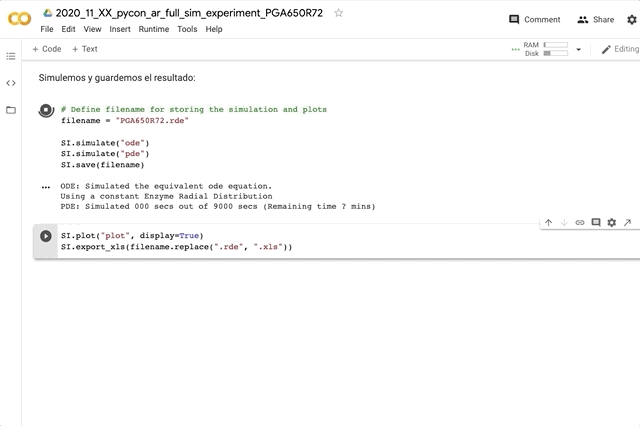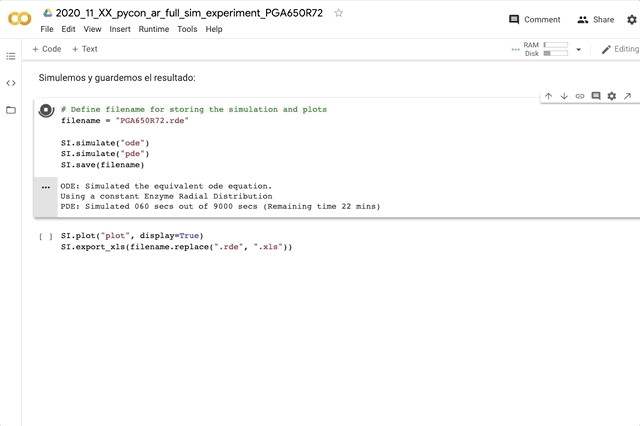
SIM2.status()
System configuration:
environment: google_colab
python version: 3.6.9
GenericSimulationLibrary version: 1.1.0
numpy version: 1.18.5
scipy version: 1.4.1
xlwt version: 1.3.0
matplotlib version: 3.2.2
dill version: 0.3.2
Inputs:
SimulationTime: 120.0
SavingTimeStep: 1.0
CatalystVolume: 0.5
BulkVolume: 100.0
Names: ('Substrat',)
InitialConcentrations: (1.3,)
EffectiveDiffusionCoefficients: (5.3e-10,)
CatalystParticleRadius: [4e-05, 6e-05, 8e-05]
CatalystParticleRadiusFrequency: [0.3, 0.5, 0.2]
ReactionFunction: <function MichaelisMenten at 0x134d50dd0>
ReactionParameters: (41, 0.13)
CatalystEnzymeConcentration: 0.35
Plot Options:
title: Simulación de Michaelis Menten para la PyconAr
label_x: Tiempo de reacción [s]
label_y: Concentración [mM]
ode_kwargs: {'label': 'ode', 'color': 'black', 'marker': '', 'markersize': 6, 'linestyle': 'dashed', 'linewidth': 2}
pde_kwargs: {'label': 'pde', 'color': 'black', 'marker': '', 'markersize': 6, 'linestyle': 'solid', 'linewidth': 2}
data_kwargs: {'label': 'exp', 'color': 'red', 'marker': 's', 'markersize': 6, 'linestyle': 'none', 'linewidth': 2}
data_x: [0.0, 30, 60, 90, 120]
data_y: [1.3, 0.65, 0.25, 0.1, 0.0]
Simulations:
ODE: yes
PDE: yes
SIM2.plot(figsize=(20,12))
Coding as a Service: librería pypsdier, aprendizajes y metodología¶
Framework Propuesto¶
La librería pypsdier es específica al problema de reacción-difusión para reactores de enzima inmobilizada, con flexibilidad para diferentes modelos de reacción y condiciones de operación.
Sin embargo, se propone un framework de trabajo con las funcionalidades mínimas: GenericSimulationLibrary
Disponible en https://github.com/sebastiandres/GenericSimulationLibrary:
- En
simulation_interface.pyactualizar los métodos: new, load, simulate, save, plot, export. - En
docs/source/*.rstactualizar la documentación.
Aprendizaje:
Puedes usar las mismas herramientas e ideas, aunque tu equipo y proyecto sea más pequeño e informal, porque simplifican y mejoran la colaboración.
- git + github/bitbucket.
- readthedocs
- pypi
- google colab/mybinder
- Interface OO