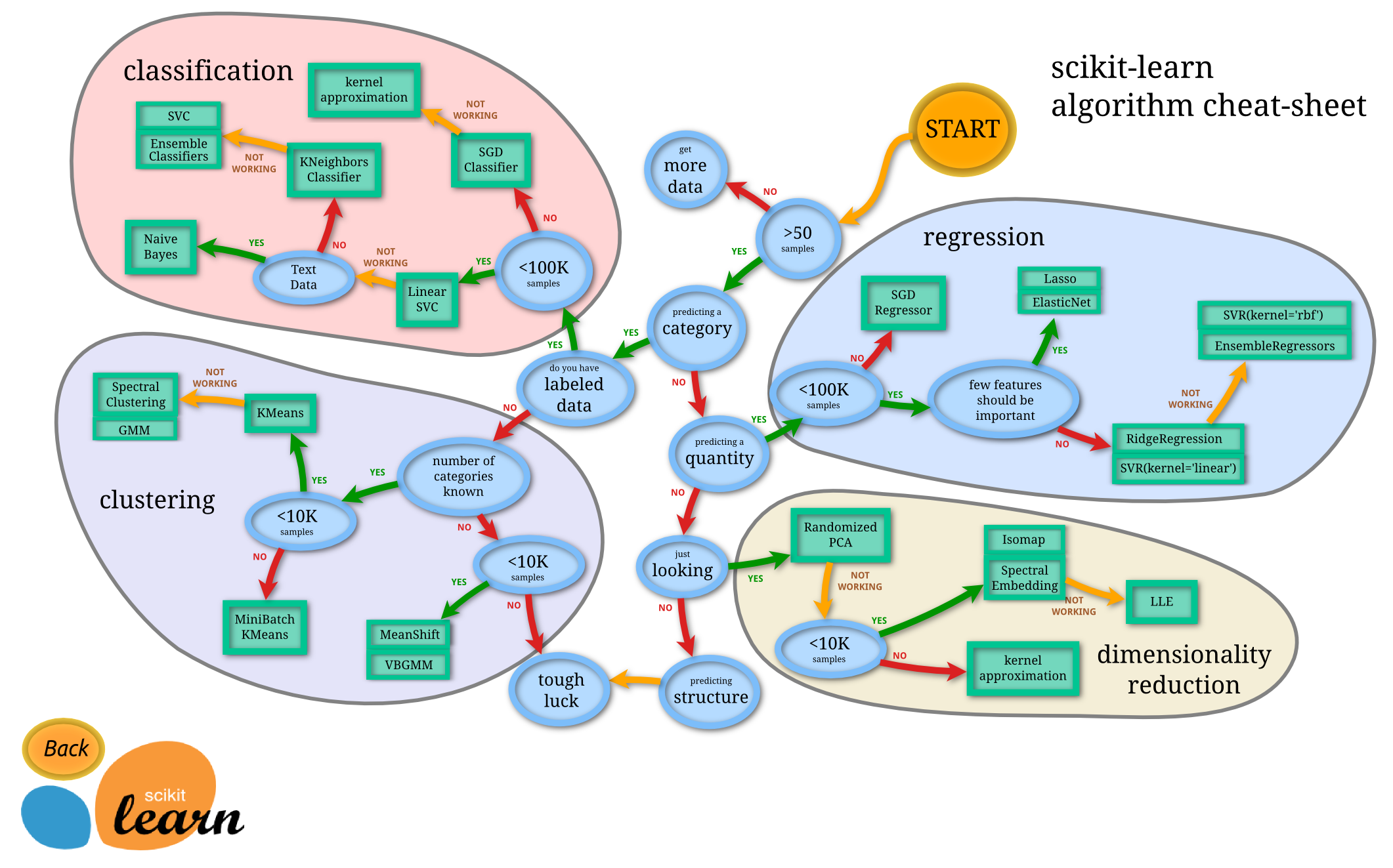
1.- Sobre la librería sklearn¶
¿Porqué sklearn?¶
sklearn viene de scientific toolbox for Machine Learning.
scikit learn para los amigos.
Existen múltiples scikits, que son "scientific toolboxes" construidos sobre SciPy: https://scikits.appspot.com/scikits.
Primero que nada... ¿Qué es Machine Learning?
2.- Machine Learning 101¶
Ejemplo¶
Consideremos un dataset consistente en características de diversos animales.
patas, ancho, largo, alto, peso, especie
[numero],[metros],[metros],[metros],[kilogramos],[]
2, 0.6, 0.4, 1.7, 75, humano
2, 0.6, 0.4, 1.8, 90, humano
...
2, 0.5, 0.5, 1.7, 85, humano
4, 0.2, 0.5, 0,3, 30, gato
...
4, 0.25, 0.55, 0.32, 32, gato
4, 0.5, 0.8, 0.3, 50, perro
...
4, 0.4, 0.4, 0.32, 40, perrofrom sklearn import HelpfulMethods
from sklearn import AlgorithmIWantToUse
# split data into train and test datasets
# train model with train dataset
# compute error on test dataset
# Optional: Train model with all available data
# Use model for some prediction
--------------------------------------------------------------------------- ImportError Traceback (most recent call last) <ipython-input-3-d2a696111fd2> in <module> ----> 1 from sklearn import HelpfulMethods 2 from sklearn import AlgorithmIWantToUse 3 4 # split data into train and test datasets 5 ImportError: cannot import name 'HelpfulMethods' from 'sklearn' (/miniconda3/envs/meetup/lib/python3.9/site-packages/sklearn/__init__.py)
4- Clustering con sklearn¶
Wine Dataset¶
Los datos del Wine Dataset son un conjunto de datos clásicos para verificar los algoritmos de clustering.

Los datos corresponden a 3 cultivos diferentes de vinos de la misma región de Italia, y que han sido identificados con las etiquetas 1, 2 y 3.
4- Clustering con sklearn¶
Wine Dataset¶
Para cada tipo de vino se realizado 13 análisis químicos:
- Alcohol
- Malic acid
- Ash
- Alcalinity of ash
- Magnesium
- Total phenols
- Flavanoids
- Nonflavanoid phenols
- Proanthocyanins
- Color intensity
- Hue
- OD280/OD315 of diluted wines
- Proline
La base de datos contiene 178 muestras distintas en total.
%%bash
head data/wine_data.csv
class,alcohol,malic_acid,ash,alcalinity_of_ash,magnesium,total_phenols,flavanoids,nonflavanoid_phenols,proanthocyanins,color_intensity,hue,OD280-OD315_of_diluted_wines,proline 1,14.23,1.71,2.43,15.6,127,2.8,3.06,.28,2.29,5.64,1.04,3.92,1065 1,13.2,1.78,2.14,11.2,100,2.65,2.76,.26,1.28,4.38,1.05,3.4,1050 1,13.16,2.36,2.67,18.6,101,2.8,3.24,.3,2.81,5.68,1.03,3.17,1185 1,14.37,1.95,2.5,16.8,113,3.85,3.49,.24,2.18,7.8,.86,3.45,1480 1,13.24,2.59,2.87,21,118,2.8,2.69,.39,1.82,4.32,1.04,2.93,735 1,14.2,1.76,2.45,15.2,112,3.27,3.39,.34,1.97,6.75,1.05,2.85,1450 1,14.39,1.87,2.45,14.6,96,2.5,2.52,.3,1.98,5.25,1.02,3.58,1290 1,14.06,2.15,2.61,17.6,121,2.6,2.51,.31,1.25,5.05,1.06,3.58,1295 1,14.83,1.64,2.17,14,97,2.8,2.98,.29,1.98,5.2,1.08,2.85,1045
import pandas as pd
data = pd.read_csv("data/wine_data.csv")
data
| class | alcohol | malic_acid | ash | alcalinity_of_ash | magnesium | total_phenols | flavanoids | nonflavanoid_phenols | proanthocyanins | color_intensity | hue | OD280-OD315_of_diluted_wines | proline | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 1 | 14.23 | 1.71 | 2.43 | 15.6 | 127 | 2.80 | 3.06 | 0.28 | 2.29 | 5.64 | 1.04 | 3.92 | 1065 |
| 1 | 1 | 13.20 | 1.78 | 2.14 | 11.2 | 100 | 2.65 | 2.76 | 0.26 | 1.28 | 4.38 | 1.05 | 3.40 | 1050 |
| 2 | 1 | 13.16 | 2.36 | 2.67 | 18.6 | 101 | 2.80 | 3.24 | 0.30 | 2.81 | 5.68 | 1.03 | 3.17 | 1185 |
| 3 | 1 | 14.37 | 1.95 | 2.50 | 16.8 | 113 | 3.85 | 3.49 | 0.24 | 2.18 | 7.80 | 0.86 | 3.45 | 1480 |
| 4 | 1 | 13.24 | 2.59 | 2.87 | 21.0 | 118 | 2.80 | 2.69 | 0.39 | 1.82 | 4.32 | 1.04 | 2.93 | 735 |
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 173 | 3 | 13.71 | 5.65 | 2.45 | 20.5 | 95 | 1.68 | 0.61 | 0.52 | 1.06 | 7.70 | 0.64 | 1.74 | 740 |
| 174 | 3 | 13.40 | 3.91 | 2.48 | 23.0 | 102 | 1.80 | 0.75 | 0.43 | 1.41 | 7.30 | 0.70 | 1.56 | 750 |
| 175 | 3 | 13.27 | 4.28 | 2.26 | 20.0 | 120 | 1.59 | 0.69 | 0.43 | 1.35 | 10.20 | 0.59 | 1.56 | 835 |
| 176 | 3 | 13.17 | 2.59 | 2.37 | 20.0 | 120 | 1.65 | 0.68 | 0.53 | 1.46 | 9.30 | 0.60 | 1.62 | 840 |
| 177 | 3 | 14.13 | 4.10 | 2.74 | 24.5 | 96 | 2.05 | 0.76 | 0.56 | 1.35 | 9.20 | 0.61 | 1.60 | 560 |
178 rows × 14 columns
data.columns
Index(['class', 'alcohol', 'malic_acid', 'ash', 'alcalinity_of_ash',
'magnesium', 'total_phenols', 'flavanoids', 'nonflavanoid_phenols',
'proanthocyanins', 'color_intensity', 'hue',
'OD280-OD315_of_diluted_wines', 'proline '],
dtype='object')
data["class"].value_counts()
2 71 1 59 3 48 Name: class, dtype: int64
data.describe(include="all")
| class | alcohol | malic_acid | ash | alcalinity_of_ash | magnesium | total_phenols | flavanoids | nonflavanoid_phenols | proanthocyanins | color_intensity | hue | OD280-OD315_of_diluted_wines | proline | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| count | 178.000000 | 178.000000 | 178.000000 | 178.000000 | 178.000000 | 178.000000 | 178.000000 | 178.000000 | 178.000000 | 178.000000 | 178.000000 | 178.000000 | 178.000000 | 178.000000 |
| mean | 1.938202 | 13.000618 | 2.336348 | 2.366517 | 19.494944 | 99.741573 | 2.295112 | 2.029270 | 0.361854 | 1.590899 | 5.058090 | 0.957449 | 2.611685 | 746.893258 |
| std | 0.775035 | 0.811827 | 1.117146 | 0.274344 | 3.339564 | 14.282484 | 0.625851 | 0.998859 | 0.124453 | 0.572359 | 2.318286 | 0.228572 | 0.709990 | 314.907474 |
| min | 1.000000 | 11.030000 | 0.740000 | 1.360000 | 10.600000 | 70.000000 | 0.980000 | 0.340000 | 0.130000 | 0.410000 | 1.280000 | 0.480000 | 1.270000 | 278.000000 |
| 25% | 1.000000 | 12.362500 | 1.602500 | 2.210000 | 17.200000 | 88.000000 | 1.742500 | 1.205000 | 0.270000 | 1.250000 | 3.220000 | 0.782500 | 1.937500 | 500.500000 |
| 50% | 2.000000 | 13.050000 | 1.865000 | 2.360000 | 19.500000 | 98.000000 | 2.355000 | 2.135000 | 0.340000 | 1.555000 | 4.690000 | 0.965000 | 2.780000 | 673.500000 |
| 75% | 3.000000 | 13.677500 | 3.082500 | 2.557500 | 21.500000 | 107.000000 | 2.800000 | 2.875000 | 0.437500 | 1.950000 | 6.200000 | 1.120000 | 3.170000 | 985.000000 |
| max | 3.000000 | 14.830000 | 5.800000 | 3.230000 | 30.000000 | 162.000000 | 3.880000 | 5.080000 | 0.660000 | 3.580000 | 13.000000 | 1.710000 | 4.000000 | 1680.000000 |
from matplotlib import pyplot as plt
data.hist(figsize=(12,20))
plt.show()
from matplotlib import pyplot as plt
#pd.scatter_matrix(data, figsize=(12,12), range_padding=0.2)
#plt.show()
X = data.drop("class", axis=1)
true_labels = data["class"] -1 # labels deben ser 0, 1, 2, ..., n-1
print(X.mean())
alcohol 13.000618 malic_acid 2.336348 ash 2.366517 alcalinity_of_ash 19.494944 magnesium 99.741573 total_phenols 2.295112 flavanoids 2.029270 nonflavanoid_phenols 0.361854 proanthocyanins 1.590899 color_intensity 5.058090 hue 0.957449 OD280-OD315_of_diluted_wines 2.611685 proline 746.893258 dtype: float64
print(X.std())
alcohol 0.811827 malic_acid 1.117146 ash 0.274344 alcalinity_of_ash 3.339564 magnesium 14.282484 total_phenols 0.625851 flavanoids 0.998859 nonflavanoid_phenols 0.124453 proanthocyanins 0.572359 color_intensity 2.318286 hue 0.228572 OD280-OD315_of_diluted_wines 0.709990 proline 314.907474 dtype: float64
from sklearn.cluster import KMeans
from sklearn.metrics import confusion_matrix
# Parameters
n_clusters = 3
# Running the algorithm
kmeans = KMeans(n_clusters)
kmeans.fit(X)
pred_labels = kmeans.labels_
cm = confusion_matrix(true_labels, pred_labels)
print(cm)
[[ 0 46 13] [50 1 20] [19 0 29]]
from sklearn import preprocessing
X_scaled = preprocessing.scale(X)
print(X_scaled.mean())
-1.2282501914608474e-16
print(X_scaled.std())
1.0
from sklearn.cluster import KMeans
from sklearn.metrics import confusion_matrix
# Parameters
n_clusters = 3
# Running the algorithm
kmeans = KMeans(n_clusters)
kmeans.fit(X_scaled)
pred_labels = kmeans.labels_
cm = confusion_matrix(true_labels, pred_labels)
print(cm)
[[ 0 59 0] [ 3 3 65] [48 0 0]]
from sklearn.cluster import KMeans
clusters = range(2,20)
total_distance = []
for n_clusters in clusters:
kmeans = KMeans(n_clusters)
kmeans.fit(X_scaled)
pred_labels = kmeans.labels_
centroids = kmeans.cluster_centers_
# Get the distances
distance_for_n = 0
for k in range(n_clusters):
points = X_scaled[pred_labels==k]
aux = (points - centroids[k,:])**2
distance_for_n += (aux.sum(axis=1)**0.5).sum()
total_distance.append(distance_for_n)
4- Clustering con sklearn¶
Graficando lo anterior, obtenemos
from matplotlib import pyplot as plt
fig = plt.figure(figsize=(16,8))
plt.plot(clusters, total_distance, 'rs')
plt.xlim(min(clusters)-1, max(clusters)+1)
plt.ylim(0, max(total_distance)*1.1)
plt.show()
4- Clustering con sklearn¶
¿Qué tan dificil es usar otro algoritmo de clustering?
Nada dificil.
Algoritmos disponibles:
- K-Means
- Mini-batch K-means
- Affinity propagation
- Mean-shift
- Spectral clustering
- Ward hierarchical clustering
- Agglomerative clustering
- DBSCAN
- Gaussian mixtures
- Birch
Lista con detalles: http://scikit-learn.org/stable/modules/clustering.html
from sklearn.cluster import KMeans
from sklearn.metrics import confusion_matrix
from sklearn import preprocessing
# Normalization of data
X_scaled = preprocessing.scale(X)
# Running the algorithm
kmeans = KMeans(n_clusters=3)
kmeans.fit(X_scaled)
pred_labels = kmeans.labels_
# Evaluating the output
cm = confusion_matrix(true_labels, pred_labels)
print(cm)
[[ 0 59 0] [ 3 3 65] [48 0 0]]
from sklearn.cluster import MiniBatchKMeans
from sklearn.metrics import confusion_matrix
from sklearn import preprocessing
# Normalization of data
X_scaled = preprocessing.scale(X)
# Running the algorithm
kmeans = MiniBatchKMeans(n_clusters=3)
kmeans.fit(X_scaled)
pred_labels = kmeans.labels_
# Evaluating the output
cm = confusion_matrix(true_labels, pred_labels)
print(cm)
[[ 0 0 59] [ 2 65 4] [48 0 0]]
from sklearn.cluster import AffinityPropagation
from sklearn.metrics import confusion_matrix
from sklearn import preprocessing
# Normalization of data
X_scaled = preprocessing.scale(X)
# Running the algorithm
kmeans = AffinityPropagation(preference=-300)
kmeans.fit(X_scaled)
pred_labels = kmeans.labels_
# Evaluating the output
cm = confusion_matrix(true_labels, pred_labels)
print(cm)
[[49 10 0] [ 3 58 10] [ 2 0 46]]
/miniconda3/envs/meetup/lib/python3.9/site-packages/sklearn/cluster/_affinity_propagation.py:148: FutureWarning: 'random_state' has been introduced in 0.23. It will be set to None starting from 1.0 (renaming of 0.25) which means that results will differ at every function call. Set 'random_state' to None to silence this warning, or to 0 to keep the behavior of versions <0.23. warnings.warn(
5- Clasificación¶
Reconocimiento de dígitos¶
Los datos se encuentran en 2 archivos, data/optdigits.train y data/optdigits.test.
Como su nombre lo indica, el set data/optdigits.train contiene los ejemplos que deben ser usados para entrenar el modelo, mientras que el set data/optdigits.test se utilizará para obtener una estimación del error de predicción.
Ambos archivos comparten el mismo formato: cada línea contiene 65 valores. Los 64 primeros corresponden a la representación de la imagen en escala de grises (0-blanco, 255-negro), y el valor 65 corresponde al dígito de la imágen (0-9).
5- Clasificación¶
Cargando los datos¶
Para cargar los datos, utilizamos np.loadtxt con los parámetros extra delimiter (para indicar que el separador será en esta ocasión una coma) y con el dype np.int8 (para que su representación en memoria sea la mínima posible, 8 bits en vez de 32/64 bits para un float).
import numpy as np
XY_tv = np.loadtxt("data/optdigits.train", delimiter=",", dtype=np.int8)
print(XY_tv)
X_tv = XY_tv[:,:64]
Y_tv = XY_tv[:, 64]
print(X_tv.shape)
print(Y_tv.shape)
print(X_tv[0,:])
print(X_tv[0,:].reshape(8,8))
print(Y_tv[0])
[[ 0 1 6 ... 0 0 0] [ 0 0 10 ... 0 0 0] [ 0 0 8 ... 0 0 7] ... [ 0 0 3 ... 0 0 6] [ 0 0 6 ... 5 0 6] [ 0 0 2 ... 0 0 7]] (3823, 64) (3823,) [ 0 1 6 15 12 1 0 0 0 7 16 6 6 10 0 0 0 8 16 2 0 11 2 0 0 5 16 3 0 5 7 0 0 7 13 3 0 8 7 0 0 4 12 0 1 13 5 0 0 0 14 9 15 9 0 0 0 0 6 14 7 1 0 0] [[ 0 1 6 15 12 1 0 0] [ 0 7 16 6 6 10 0 0] [ 0 8 16 2 0 11 2 0] [ 0 5 16 3 0 5 7 0] [ 0 7 13 3 0 8 7 0] [ 0 4 12 0 1 13 5 0] [ 0 0 14 9 15 9 0 0] [ 0 0 6 14 7 1 0 0]] 0
5- Clasificación¶
Visualizando los datos¶
Para visualizar los datos utilizaremos el método imshow de pyplot. Resulta necesario convertir el arreglo desde las dimensiones (1,64) a (8,8) para que la imagen sea cuadrada y pueda distinguirse el dígito. Superpondremos además el label correspondiente al dígito, mediante el método text. Realizaremos lo anterior para los primeros 25 datos del archivo.
from matplotlib import pyplot as plt
# Well plot the first nx*ny examples
nx, ny = 5, 5
fig, ax = plt.subplots(nx, ny, figsize=(12,12))
for i in range(nx):
for j in range(ny):
index = j+ny*i
data = X_tv[index,:].reshape(8,8)
label = Y_tv[index]
ax[i][j].imshow(data, interpolation='nearest', cmap=plt.get_cmap('gray_r'))
ax[i][j].text(7, 0, str(int(label)), horizontalalignment='center',
verticalalignment='center', fontsize=10, color='blue')
ax[i][j].get_xaxis().set_visible(False)
ax[i][j].get_yaxis().set_visible(False)
plt.show()
from sklearn.neighbors import KNeighborsClassifier
k = 1
kNN = KNeighborsClassifier(n_neighbors=k)
kNN.fit(X_tv, Y_tv)
Y_pred = kNN.predict(X_tv)
n_errors = sum(Y_pred!=Y_tv)
print("Hay %d errores de un total de %d ejemplos de entrenamiento" %(n_errors, len(Y_tv)))
Hay 0 errores de un total de 3823 ejemplos de entrenamiento
¡La mejor predicción del punto es el mismo punto!
Pero esto generalizaría catastróficamente.
Es importantísimo entrenar en un set de datos y luego probar como generaliza/funciona en un set completamente nuevo.
5- Clasificación¶
Seleccionando el número adecuado de vecinos¶
Buscando el valor de k más apropiado
A partir del análisis del punto anterior, nos damos cuenta de la necesidad de:
- Calcular el error en un set distinto al utilizado para entrenar.
- Calcular el mejor valor de vecinos para el algoritmo.
(Esto tomará un tiempo)
from sklearn.neighbors import KNeighborsClassifier
from sklearn.model_selection import train_test_split
template = "k={0:,d}: {1:.1f} +- {2:.1f} errores de clasificación de un total de {3:,d} puntos"
# Fitting the model
mean_error_for_k = []
std_error_for_k = []
k_range = range(1,8)
for k in k_range:
errors_k = []
for i in range(10):
kNN = KNeighborsClassifier(n_neighbors=k)
X_train, X_valid, Y_train, Y_valid = train_test_split(X_tv, Y_tv, train_size=0.75)
kNN.fit(X_train, Y_train)
# Predicting values
Y_valid_pred = kNN.predict(X_valid)
# Count the errors
n_errors = sum(Y_valid!=Y_valid_pred)
# Add them to vector
errors_k.append(100.*n_errors/len(Y_valid))
errors = np.array(errors_k)
print(template.format(k, errors.mean(), errors.std(), len(Y_valid)))
mean_error_for_k.append(errors.mean())
std_error_for_k.append(errors.std())
k=1: 1.6 +- 0.3 errores de clasificación de un total de 956 puntos k=2: 2.3 +- 0.5 errores de clasificación de un total de 956 puntos k=3: 1.6 +- 0.3 errores de clasificación de un total de 956 puntos k=4: 2.0 +- 0.4 errores de clasificación de un total de 956 puntos k=5: 1.7 +- 0.2 errores de clasificación de un total de 956 puntos k=6: 1.8 +- 0.3 errores de clasificación de un total de 956 puntos k=7: 1.7 +- 0.4 errores de clasificación de un total de 956 puntos
5- Clasificación¶
Podemos visualizar los datos anteriores utilizando el siguiente código, que requiere que sd_error_for k y mean_error_for_k hayan sido apropiadamente definidos.
mean = np.array(mean_error_for_k)
std = np.array(std_error_for_k)
plt.figure(figsize=(12,8))
plt.plot(k_range, mean - std, "k:")
plt.plot(k_range, mean , "r.-")
plt.plot(k_range, mean + std, "k:")
plt.xlabel("Numero de vecinos k")
plt.ylabel("Error de clasificacion")
plt.show()
from sklearn.neighbors import KNeighborsClassifier
from sklearn.model_selection import train_test_split
import numpy as np
k = 3
kNN = KNeighborsClassifier(n_neighbors=k)
kNN.fit(X_tv, Y_tv)
KNeighborsClassifier(n_neighbors=3)
# Cargando el archivo data/optdigits.tes
XY_test = np.loadtxt("data/optdigits.test", delimiter=",")
X_test = XY_test[:,:64]
Y_test = XY_test[:, 64]
# Predicción de etiquetas
Y_pred = kNN.predict(X_test)
5- Clasificación¶
Puesto que tenemos las etiquetas verdaderas en el set de entrenamiento, podemos visualizar que números han sido correctamente etiquetados.
from matplotlib import pyplot as plt
# Mostrar los datos correctos
mask = (Y_pred==Y_test)
X_aux = X_test[mask]
Y_aux_true = Y_test[mask]
Y_aux_pred = Y_pred[mask]
# We'll plot the first 100 examples, randomly choosen
nx, ny = 5, 5
fig, ax = plt.subplots(nx, ny, figsize=(12,12))
for i in range(nx):
for j in range(ny):
index = j+ny*i
data = X_aux[index,:].reshape(8,8)
label_pred = str(int(Y_aux_pred[index]))
label_true = str(int(Y_aux_true[index]))
ax[i][j].imshow(data, interpolation='nearest', cmap=plt.get_cmap('gray_r'))
ax[i][j].text(0, 0, label_pred, horizontalalignment='center',
verticalalignment='center', fontsize=10, color='green')
ax[i][j].text(7, 0, label_true, horizontalalignment='center',
verticalalignment='center', fontsize=10, color='blue')
ax[i][j].get_xaxis().set_visible(False)
ax[i][j].get_yaxis().set_visible(False)
plt.show()
from matplotlib import pyplot as plt
# Mostrar los datos correctos
mask = (Y_pred!=Y_test)
X_aux = X_test[mask]
Y_aux_true = Y_test[mask]
Y_aux_pred = Y_pred[mask]
# We'll plot the first 100 examples, randomly choosen
nx, ny = 5, 5
fig, ax = plt.subplots(nx, ny, figsize=(12,12))
for i in range(nx):
for j in range(ny):
index = j+ny*i
data = X_aux[index,:].reshape(8,8)
label_pred = str(int(Y_aux_pred[index]))
label_true = str(int(Y_aux_true[index]))
ax[i][j].imshow(data, interpolation='nearest', cmap=plt.get_cmap('gray_r'))
ax[i][j].text(0, 0, label_pred, horizontalalignment='center',
verticalalignment='center', fontsize=10, color='red')
ax[i][j].text(7, 0, label_true, horizontalalignment='center',
verticalalignment='center', fontsize=10, color='blue')
ax[i][j].get_xaxis().set_visible(False)
ax[i][j].get_yaxis().set_visible(False)
plt.show()
# Error global
mask = (Y_pred!=Y_test)
error_prediccion = 100.*sum(mask) / len(mask)
print("Error de predicción total de {0:.1f} %".format(error_prediccion))
for digito in range(0,10):
mask_digito = Y_test==digito
Y_test_digito = Y_test[mask_digito]
Y_pred_digito = Y_pred[mask_digito]
mask = Y_test_digito!=Y_pred_digito
error_prediccion = 100.*sum((Y_pred_digito!=Y_test_digito)) / len(Y_pred_digito)
print("Error de predicción para digito {0:d} de {1:.1f} %".format(digito, error_prediccion))
Error de predicción total de 2.2 % Error de predicción para digito 0 de 0.0 % Error de predicción para digito 1 de 1.1 % Error de predicción para digito 2 de 2.3 % Error de predicción para digito 3 de 1.1 % Error de predicción para digito 4 de 1.7 % Error de predicción para digito 5 de 1.6 % Error de predicción para digito 6 de 0.0 % Error de predicción para digito 7 de 3.9 % Error de predicción para digito 8 de 6.9 % Error de predicción para digito 9 de 3.3 %
from sklearn.metrics import confusion_matrix as cm
cm = cm(Y_test, Y_pred)
print(cm)
[[178 0 0 0 0 0 0 0 0 0] [ 0 180 0 0 0 0 1 0 1 0] [ 0 4 173 0 0 0 0 0 0 0] [ 0 0 0 181 0 0 0 1 1 0] [ 0 2 0 0 178 0 0 0 1 0] [ 0 0 0 1 1 179 0 0 0 1] [ 0 0 0 0 0 0 181 0 0 0] [ 0 0 0 0 0 0 0 172 1 6] [ 0 9 0 1 0 0 0 0 162 2] [ 0 0 0 4 0 1 0 0 1 174]]
# As in http://scikit-learn.org/stable/auto_examples/model_selection/plot_confusion_matrix.html
def plot_confusion_matrix(cm, title='Confusion matrix', cmap=plt.cm.jet):
plt.figure(figsize=(10,10))
plt.imshow(cm, interpolation='nearest', cmap=cmap)
plt.title(title)
plt.colorbar()
tick_marks = np.arange(10)
plt.xticks(tick_marks, tick_marks)
plt.yticks(tick_marks, tick_marks)
plt.tight_layout()
plt.ylabel('True label')
plt.xlabel('Predicted label')
plt.show()
return None
# Compute confusion matrix
plt.figure()
plot_confusion_matrix(cm)
<Figure size 432x288 with 0 Axes>
# Normalize the confusion matrix by row (i.e by the number of samples in each class)
cm_normalized = cm.astype('float') / cm.sum(axis=1)[:, np.newaxis]
plot_confusion_matrix(cm_normalized, title='Normalized confusion matrix')
5- Clasificación¶
A partir de lo anterior, vemos observamos que los mayores errores son:
- El 2 puede clasificarse erróneamente como 1 (pero no viceversa).
- El 7 puede clasificarse erróneamente como 9 (pero no viceversa).
- El 8 puede clasificarse erróneamente como 1 (pero no viceversa).
- El 9 puede clasificarse erróneamente como 3 (pero no viceversa).
Múltiples familias de algoritmos:
- Logistic Regression
- Naive Bayes
- Decision Trees
- Random Forests
- Support Vector Machines
- Neural Networks
- Etc etc
link: http://scikit-learn.org/stable/supervised_learning.html
5- Conclusión¶
Sklearn tiene muchos algoritmos implementados y es fácil de usar.
Sin embargo, hay que tener presente GIGO: Garbage In, Garbage Out:
- Exploración y visualización inicial de datos.
- Limpieza de datos
- Utilización del algoritmo requiere conocer su funconamiento para mejor tuneo de parámetros.
- Es bueno y fácil probar más de un algoritmo.
5- Conclusión¶
Y por último:
- Aplicación de algoritmos de ML es delicado porque requiere (1) conocer bien los datos y (2) entender las limitaciones del algoritmo.
- Considerar siempre una muestra para entrenamiento y una muestra para testeo: predicción es inútil si no se entrega un margen de error para la predicción.